2024年5月20日,北京大学现代农业研究院郭立研究员团队与何航研究员团队合作在国际著名期刊《Nature Communications》上在线发表了题为“Two telomere-to-telomere gapless genomes reveal insights into Capsicum evolution and capsaicinoid biosynthesis”的研究论文。该研究通过系统基因组分析,估计了辣椒素生物合成的进化时间,以及允许组织特异性生物合成基因协同调节和辣椒素积累。这些T2T基因组资源将加速辣椒的遗传改良,有助于了解辣椒基因组的进化。

导读
辣椒(Capsicum)因其独特的果实辛辣而闻名,这是由于辣椒素的存在。由于缺乏高质量的辣椒基因组,辣椒素生物合成的进化历史及其组织特异性机制仍不清楚。这篇文章研究人员通过组装两个端粒到端粒(T2T)无缺口辣椒基因组:一年生栽培辣椒(C.annuum)和不产辣椒素的野生辣椒(C.rhomboideum),深入解析了辣椒着丝粒序列特征,发现独特的重复序列元件;通过对辣椒素基因组的分析,研究者估计了辣椒素生物合成的进化时间。揭示了非辛辣物种中关键生物合成基因的编码和调控区被破坏。研究人员还发现了保守的胎盘特异性染色质区域,这可能允许组织特异性生物合成基因的协同调节和辣椒素积累。这些T2T基因组资源将加速辣椒的遗传改良,有助于理解辣椒基因组的进化。

T2T无缺口辣椒基因组组装
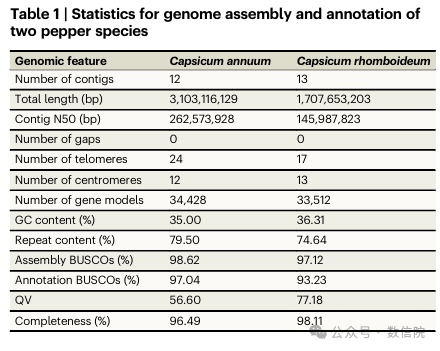
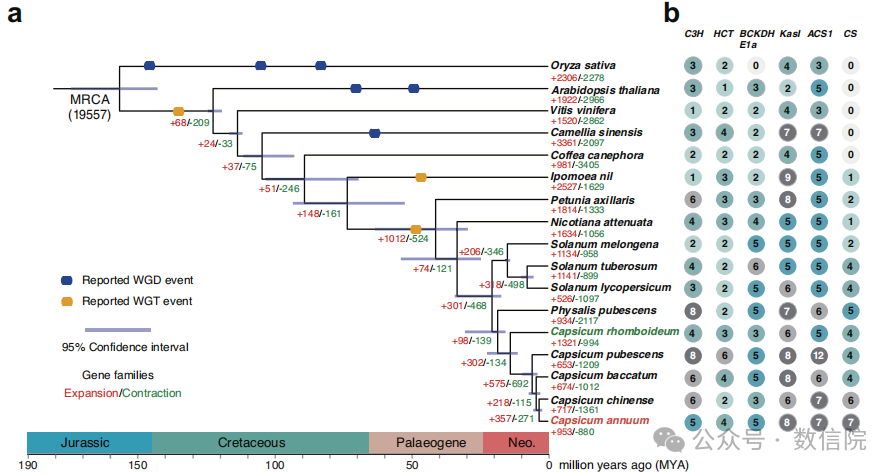
为了获得产生辣椒素的C. annuum和不产生辣椒素的C. rhomboideum的端粒到端粒的、无缺口的基因组组装结果,作者利用PacBio HiFi、ONT、Illumina和Hi-C综合技术对辛辣辣椒栽培品种(C. annuum, Ca59)和非辛辣辣椒野生品种(C. rhomboideum)进行了测序。首先使用hifiasm 来组装HiFi reads和使用NextDenovo来组装ONT reads。紧接着该研究使用ONT组装来填补HiFi组装中的缺口并修补HiFi组装中的端粒,为确保组装结果的准确性,将ONT来源的序列替换为相应的HiFi组装contigs,然后通过Hi-C挂载到12条染色体(图1a),并使用Juicebox进行手动纠正组装的错误部分。除了rDNA阵列和端粒修补,最终得到了两个辣椒的T2T无缺口基因组。C. annuum(CaT2T)包含12条染色体,基因组大小为3.1 Gb,contig N50为262.6 Mb;C.annuum基因组组装代表了迄今为止报道的最大的完整基因组序列 ,C.rhomboideum(CrT2T)包含13条染色体,基因组大小为1.7 Gb ,contig N50为146.0 Mb,代表了非驯化辣椒的无gap基因组。作者鉴定了C. annuum中完整的端粒集(24/24)和C. rhomboideum中大多数的端粒(17/26)。共线性分析显示,45.07%的C. rhomboideum基因与C. annuum具有共线性,但它们的全基因组比对显示出低序列一致性,表明存在重大分歧。通过比较两个基因组,我们证明了至少需要 10 次断裂和 11 次染色体融合才能从C. rhomboideum的核型到C. annuum的核型。
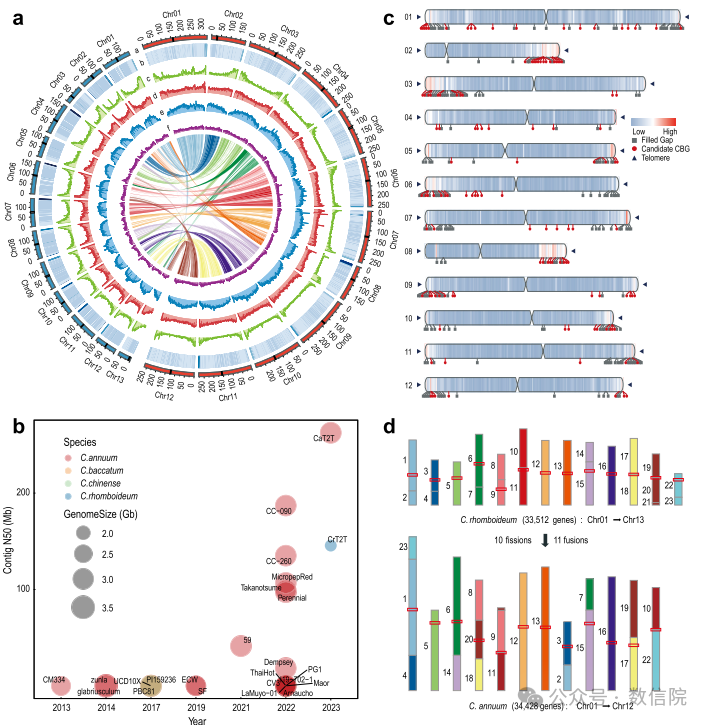
基因组验证和注释
人员对两个T2T基因组装配体进行了广泛的验证。检查了它们的Hi-C染色质相互作用图,结果显示在CaT2T和CrT2T组装体中没有明显的重叠群错位。CaT2T和CrT2T的QV分别为56.60和77.18,BUSCO评分分别为98.62%和97.12%,证明了两种组装件的高准确度和完整性(表1)。此外, Ca59与CaT2T表现出很强的共线性。此外,在 CaT2T 和 CrT2T 中观察到的对应于线粒体或叶绿体基因组的映射序列,表明最近存在质体基因组向核基因组中的整合。
重复注释表明,79.5%(2.45Gb)和74.6%(1.28Gb)的C.annuum和C.Rhomboideum基因组是重复序列,主要由转座因子(TE),特别是长末端重复(LTR)逆转录转座子组成。而LTR插入在C.rhomboideum的出现相对较晚,但C. annuum在大约 10 万年前(Mya)和 3.9 Mya 有两次插入的爆发,这与之前关于Ca59组装的报告一致。两种辣椒基因组的卫星重复序列含量均较低(<0.01%),远低于人类(4.5%)、模式植物拟南芥(0.37%)及茄科近缘种烟草(1.75%)。通过结合从头预测、同源蛋白和转录组数据,分别预测了 CaT2T 和 CrT2T 的 34,428 个和 33,512 个蛋白质编码基因。CaT2T填补的gap编码了614个基因,其中110个是首次被注释(与Ca59非同源)。两个辣椒基因组染色体末端的基因分布密集,但在着丝粒的基因分布稀疏。CaT2T 编码 117 个推定的辣椒素类生物合成基因(CBGs),包括先前报道的编码辣椒素合酶(CS)、氨甲基转移酶(AMT)、酮酰基-ACP 合酶(Kas)和酰基载体蛋白(ACL)的基因。这些推定的CBGs在至少一个组织中表达,其中26个在果实胎座中的表达水平远高于其他组织。因此,这些基因可能是辣椒素类生物合成酶的有力候选者。
辣椒着丝粒被CRM反转录转录子广泛入侵
研究者通过CENH3 ChIP-seq数据检测了CaT2T基因组的着丝粒,CENH3 ChIP-seq数据清晰地定界了12个着丝粒的位置和边界。与拟南芥和人类基因组中的着丝粒不一样,该研究的辣椒基因组的着丝粒中缺少高拷贝的串联重复序列和高度有序的重复序列,这表明该物种的着丝粒可能是刚形成的。但是,该研究发现辣椒基因组中的着丝粒被大量的Gypsy型LTR入侵,其序列占到所有着丝粒序列的71%和所有基因组序列的47.3%-49.7%。这种模式在einkorn小麦和棉花中也被报道过,在这两个物种中有功能的着丝粒序列中80%是Gypsy型LTR。该研究还发现着丝粒的LTR爆发式入侵的时间是晚于其爆发式入侵整个基因组的,表明最近的着丝粒进化被LTR插入影响。LTRs通常会产生片段化的或单独的LTRs,这是由于不等同源重组事件打散和重排了LTRs。在两个辣椒基因组中发现了很多单独的和完整的LTRs。该研究发现在辣椒基因组的着丝粒区域,单独的LTRs与完整的LTRs的比例相较整个基因组要低,这表明其去除LTRs的能力比非着丝粒区域要弱。共线性分析发现玉米的着丝粒反转座子(CRMs)主要富集在一些辣椒基因组的着丝粒区域,表明在不依靠CHIP-seq数据的情况下,CRMs的分布可以作为识别辣椒基因组着丝粒的一种标记。CRMs具有染色质域或CR基序,可以与着丝粒组蛋白互作且在着丝粒的进化和功能方面具有重要的作用。该研究对两个辣椒基因组和马铃薯基因组的Gypsy型LTRs的系统发育分析表明,在Gypsy型LTRs的六个亚家族中,有两个主要的亚家族,分别是Athila和Tekay。Athila亚家族的LTRs是拟南芥着丝粒中的主要LTRs,而在辣椒着丝粒中,主要的LTRs是CRM Gypsy型LTRs。缺少微卫星重复以及富含CRM型LTRs这两个特征将辣椒的着丝粒与其他报道过的植物T2T基因组的着丝粒区别开来。在两个辣椒基因组中,该研究发现物种间和染色体间的低着丝粒序列一致度,表明辣椒着丝粒在物种内和物种间的快速分化,且这个结果也和最近在拟南芥着丝粒中的报道一致。

着丝粒和端粒具有转录和表观活性
在蛋白质编码基因以及转录和表观遗传控制方面,着丝粒和端粒是研究较少的的基因组区域。基因组注释显示,CaT2T着丝粒中的60个基因在冷冻反应、DNA拓扑变化和减数分裂染色体分离等功能方面具有重要作用。相比之下,CrT2T着丝粒上编码了94个基因,富含对UV-B的响应、光合作用和昼夜节律调节。有趣的是,在两个辣椒中只有6或7个基因是同源的。着丝粒基因的低同源性反映了物种之间着丝粒的高度差异。该研究还在CaT2T的着丝粒和端粒上发现转座子和蛋白质编码基因的转录活性。

辣椒素生物合成途径的进化史
通过使用两个T2T辣椒基因组和其他14个被子植物的基因组进行系统发育组学,包括3个辛辣物种和13个非辛辣物种。结果表明,Capsicum与Physalis的亲缘关系比与Solanum的亲缘关系更强,并且分别在~17 Mya和~19 Mya处与两个分类群分化。辣椒素的生物合成仅限于Capsicum这一事实表明,在辣椒在~17 Mya与Physalis分化后,辣椒中一定出现了特殊的代谢物。此外,C. baccatum在 ~ 5 Mya 处与C.annuum和C.chinense分化,后者在 ~13.4 Mya 处与非辛辣C. rhomboideum分化,表明辣椒素途径可能起源于 13.4 Mya 和 5 Mya 之间。为了了解辣椒属作物中该途径的起源,使用OrthoFinder在17个被子植物中鉴定了与已知辣椒素生物合成基因(CBG)同源的基因。在所有物种中,无论辣味与否,都含有大多数CBG的同源基因,只有最关键的基因CS仅在茄科作物中以串联重复的形式出现。一年生辣椒含有7个串联重复拷贝,而在野生辣椒中仅存在4个拷贝。转录组分析显示CBG基因在辣椒果实中高度表达,而在不辣的植物如番茄、马铃薯、酸浆和野生辣椒中CS基因几乎不表达。序列比对显示辣椒的CS基因(CS-1/CS-2)具有保守的编码序列和上下游调控区,而在不辣的辣椒和近缘茄科物种中,CS基因编码区和调控区发生了明显的结构变异,由此导致辣椒素合成能力的差异。

染色质可及性调控组织特异性的辣椒素生物合成
辣椒素的生物合成具有高度组织特异性,并且只发生在果实中,特别是在胎盘组织中,从开花后约16天开始。为了了解组织特异性是如何实现的,研究人员对栽培辣椒果实和叶片进行了多组学联合分析,包括ATAC-seq、WGBS和RNA-seq分析,并以CaT2T为参照对数据进行了分析。RNA-seq分析表明,CS及其转录调节因子MYB31和MYB48在胎盘中特异性表达。在CS-2、MYB31和MYB48上游2kb范围内检测到了低甲基化水平的胎盘特异性开放染色质区(OCR),而CS-1在果肉和种子中也出现了 OCR,这表明CS-2可能是促成胎盘组织特异性辣椒素合成的主要功能基因。
如何对基因组分散的CBGs进行核心调控,使辣椒素的产生具有这种时间-空间特异性,仍然是一个未知数。研究人员提取了通过 ATAC-seq 鉴定的胎盘相关 OCRs 和 CBGs 上游 2 kb 序列,进行了序列motif富集分析,共获得38个富集motifs。通过两次富集分析获得的重叠富集motif揭示了五个TFBS,即MYB、G-box、Box-4、ABRE和 MYC,它们存在于所有七个CBGs中,包括CS、ACL、KasI、PAL、CCoAOMT和BCAT。这些在常见OCR中明显富集的TFBS可能被某些TFs(如 MYB31)识别,这些TFs在特定组织中与这些CBGs核心配对。在无辛辣味的栽培辣椒中,CS-2的OCRs由于2.4 kb的缺失而丢失,导致这些品种缺乏辣椒素。简而言之,多组数据分析表明辣椒中辣椒素生物合成基因的组织特异性核心调控可能属于一种表观遗传机制。

总结
在这项研究中,研究者组装了迄今为止最大的完整的植物基因组组装(C.annuum)和辣椒属的两个T2T无缺口基因组。基于辣椒T2T基因组的系统基因组学和多组学研究揭示了辣椒果实中辣椒素独特的组织特异性积累的进化机制。这些T2T基因组资源是作物基因组研究的重要里程碑,将加速辣椒研究,促进精准改良。